ในกระบวนการดำเนินการศึกษาทางพันธุกรรม เรามักพบตัวอย่าง RNA ไม่เพียงพอ ตัวอย่างเช่น สำหรับการศึกษาเนื้องอกในช่องปากทางกายวิภาคขนาดเล็ก แม้แต่ตัวอย่างเซลล์เดียว และตัวอย่างการกลายพันธุ์ของยีนเฉพาะที่คัดลอกในระดับที่ต่ำมากในเซลล์ของมนุษย์แน่นอน สำหรับการทดสอบ COVID-19 หากก้อนตรวจไม่อยู่ในตำแหน่งที่ถูกต้องหรือมีเวลาไม่เพียงพอระหว่างการสุ่มตัวอย่าง ขนาดตัวอย่างจะต่ำมาก ซึ่งเป็นสาเหตุที่คณะกรรมการสุขภาพและการวางแผนครอบครัวออกมาเมื่อสองวันก่อนและ ผ่านการทดสอบแล้ว และหากเครื่องเก็บตัวอย่างกรดนิวคลีอิกเก็บตัวอย่างกรดนิวคลีอิกไม่ได้หกตัวอย่าง คุณสามารถรายงานได้
ความไวของรีเอเจนต์มีความสำคัญเนื่องจากเรามีปัญหานี้หรือปัญหานั้น ดังนั้น เราจะทำอย่างไรเพื่อปรับปรุงความไวของ RT-PCR
ก่อนที่เราจะพูดถึงวิธีแก้ปัญหาที่เป็นไปได้ ขอกล่าวถึงภาวะแทรกซ้อนใหญ่สองประการของสถานการณ์ที่เราเพิ่งกล่าวถึง
ก่อนอื่น เรากังวลเกี่ยวกับการสูญเสีย RNA เมื่อเรามีประชากรเซลล์เพียงไม่กี่ตัวในตัวอย่างของเราหากใช้วิธีการแยกและทำความสะอาดแบบดั้งเดิม เช่น วิธีคอลัมน์หรือวิธีการตกตะกอนด้วยกรดนิวคลีอิก มีความเป็นไปได้สูงที่ตัวอย่างบางส่วนจะสูญหายทางออกหนึ่งคือการเพิ่มโมเลกุลพาหะ เช่น tRNA แต่ถึงอย่างนั้น ก็ไม่มีการรับประกันว่าการทดลองการกู้คืนของเราจะใช้ได้
ดังนั้นอะไรคือวิธีที่ดีกว่า?ตัวเลือกที่ดีสำหรับเซลล์เพาะเลี้ยงหรือตัวอย่างระดับจุลภาคคือการใช้การสลายโดยตรง
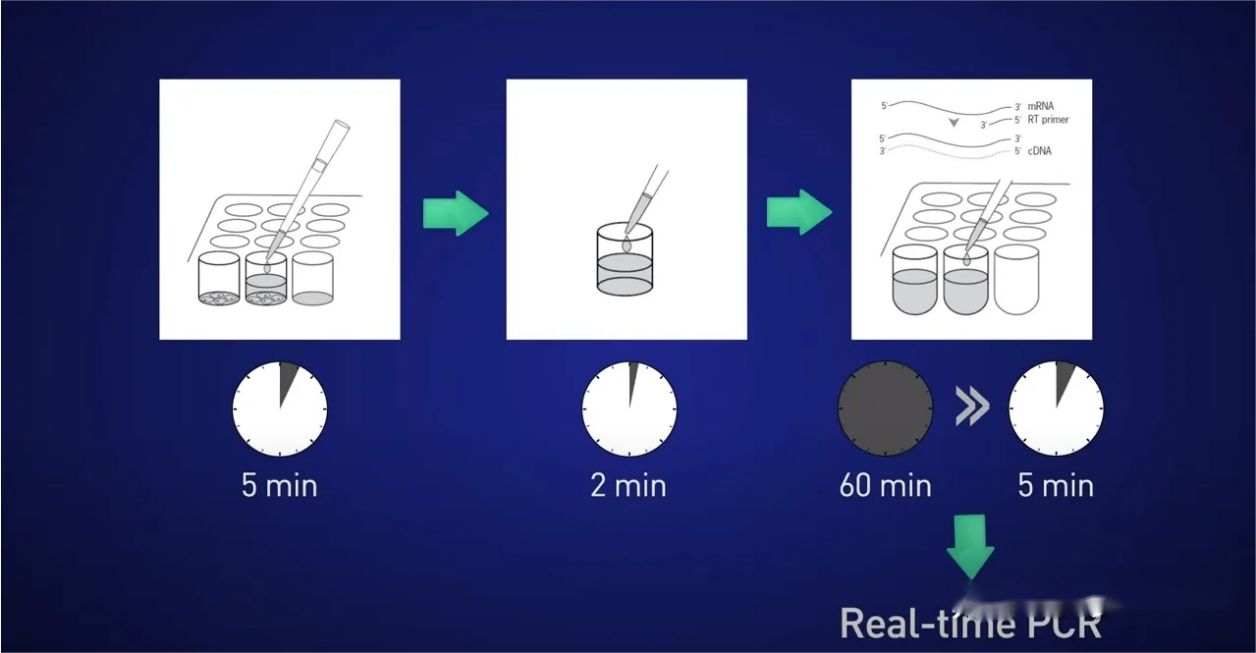
แนวคิดคือการแยกเซลล์เป็นเวลา 5 นาที ปล่อย RNA ลงในสารละลาย จากนั้นหยุดปฏิกิริยาเป็นเวลา 2 นาที จากนั้นเพิ่ม lysate ลงในปฏิกิริยาการถอดรหัสแบบย้อนกลับโดยตรง เพื่อไม่ให้ RNA สูญหาย และสุดท้าย ใส่ cDNA ที่เป็นผลลัพธ์โดยตรง สู่ปฏิกิริยาแบบเรียลไทม์
แต่จะเป็นอย่างไรถ้าเนื่องจากจุดเริ่มต้นที่จำกัดหรือการแสดงออกของยีนเป้าหมายจำนวนน้อย เราสามารถรีไซเคิล RNA ทั้งหมดและยังไม่มีเทมเพลตเพียงพอที่จะรับสัญญาณแบบเรียลไทม์ที่ดี
ในกรณีนี้ ขั้นตอนก่อนขยายจะมีประโยชน์มาก
ต่อไปนี้เป็นแผนเพื่อเพิ่มความไวหลังจากการถอดความแบบย้อนกลับก่อนเริ่มต้น เราต้องถามปลายน้ำว่าเป้าหมายใดที่เราสนใจ เพื่อที่จะออกแบบไพรเมอร์เฉพาะสำหรับเป้าหมายเหล่านี้สำหรับการขยายเสียงล่วงหน้า
สามารถทำได้โดยการสร้างไพรเมอร์ผสมที่มีไพรเมอร์มากถึง 100 คู่และรอบการเกิดปฏิกิริยา 10 ถึง 14 ครั้งดังนั้น Master Mix ที่ออกแบบมาโดยเฉพาะสำหรับข้อกำหนดนี้จึงจำเป็นในการขยาย cDNA ที่ได้รับล่วงหน้า
เหตุผลในการตั้งค่าจำนวนรอบระหว่าง 10 ถึง 14 คือจำนวนรอบที่จำกัดนี้ทำให้แน่ใจได้ว่ามีการสุ่มระหว่างเป้าหมายต่างๆ ซึ่งเป็นสิ่งสำคัญสำหรับนักวิจัยที่ต้องการข้อมูลโมเลกุลเชิงปริมาณ
หลังจากการขยายสัญญาณล่วงหน้า เราจะได้รับ cDNA จำนวนมาก ดังนั้นความไวในการตรวจจับที่ส่วนหลังจึงดีขึ้นอย่างมาก และเรายังสามารถเจือจางตัวอย่างและทำปฏิกิริยา PCR แบบเรียลไทม์หลายครั้งเพื่อกำจัดข้อผิดพลาดแบบสุ่มที่เป็นไปได้
เวลาโพสต์: 11 เม.ย.-2566